Web Uygulamamızı 20 Kat Hızlandırmak için WebAssembly'ı Nasıl Kullandık (Örnek Olay)
Yayınlanan: 2022-03-10Duymadıysanız, işte TL;DR: WebAssembly, JavaScript'in yanı sıra tarayıcıda çalışan yeni bir dildir. Evet bu doğru. JavaScript artık tarayıcıda çalışan tek dil değil!
Ancak sadece “JavaScript değil” olmanın ötesinde, ayırt edici faktörü, C/C++/Rust ( ve daha fazlası! ) gibi dillerden WebAssembly'ye kod derleyebilmeniz ve bunları tarayıcıda çalıştırabilmenizdir. WebAssembly statik olarak yazıldığından, doğrusal bir bellek kullandığından ve kompakt bir ikili biçimde depolandığından, aynı zamanda çok hızlıdır ve sonunda kodu "yerel" hızlarda, yani sizin hızınıza yakın hızlarda çalıştırmamıza izin verebilir. d ikili dosyayı komut satırında çalıştırarak elde edin. Tarayıcıda kullanım için mevcut araçlardan ve kitaplıklardan yararlanma yeteneği ve bununla bağlantılı hızlanma potansiyeli, WebAssembly'ı web için bu kadar çekici kılan iki nedendir.
Şimdiye kadar WebAssembly, oyun oynamaktan (örn. Doom 3), masaüstü uygulamalarını web'e taşımaya (örn. Autocad ve Figma) kadar her türlü uygulama için kullanıldı. Örneğin, sunucusuz bilgi işlem için verimli ve esnek bir dil olarak tarayıcının dışında bile kullanılır.
Bu makale, bir veri analizi web aracını hızlandırmak için WebAssembly kullanımına ilişkin bir vaka çalışmasıdır. Bu amaçla, aynı hesaplamaları yapan, C ile yazılmış mevcut bir aracı alıp WebAssembly'de derleyeceğiz ve yavaş JavaScript hesaplamalarını değiştirmek için kullanacağız.
Not : Bu makale, C kodu derleme gibi bazı ileri düzey konuları ele almaktadır, ancak bununla ilgili deneyiminiz yoksa endişelenmeyin; yine de takip edebilecek ve WebAssembly ile neyin mümkün olduğuna dair bir fikir edinebileceksiniz.
Arka fon
Birlikte çalışacağımız web uygulaması, bilim insanlarına DNA dizileme verilerinin kalitesinin hızlı bir ön izlemesini sağlayan etkileşimli bir web aracı olan fastq.bio; dizileme, bir DNA örneğindeki "harfleri" (yani nükleotidleri) okuduğumuz süreçtir.
Uygulamanın çalışırken bir ekran görüntüsü:
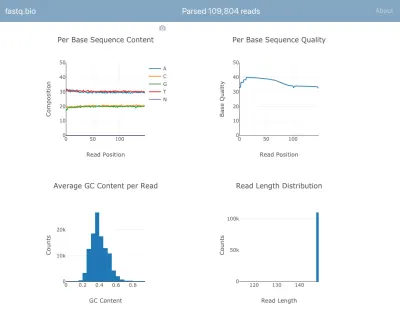
Hesaplamaların ayrıntılarına girmeyeceğiz, ancak özetle yukarıdaki grafikler, bilim insanlarına sıralamanın ne kadar iyi gittiğine dair bir fikir veriyor ve bir bakışta veri kalitesi sorunlarını belirlemek için kullanılıyor.
Bu tür kalite kontrol raporları oluşturmak için düzinelerce komut satırı aracı olmasına rağmen, fastq.bio'nun amacı, tarayıcıdan çıkmadan veri kalitesinin etkileşimli bir önizlemesini sunmaktır. Bu, özellikle komut satırı konusunda rahat olmayan bilim adamları için kullanışlıdır.
Uygulamaya giriş, dizileme aracı tarafından çıkarılan ve DNA dizilerinin bir listesini ve DNA dizilerindeki her nükleotit için bir kalite puanını içeren bir düz metin dosyasıdır. Bu dosyanın formatı “FASTQ” olarak bilinir, dolayısıyla fastq.bio adı.
FASTQ biçimini merak ediyorsanız (bu makaleyi anlamak için gerekli değildir), FASTQ için Wikipedia sayfasına bakın. (Uyarı: FASTQ dosya biçiminin yüz avuçlarını tetiklediği sahada bilinmektedir.)
fastq.bio: JavaScript Uygulaması
fastq.bio'nun orijinal sürümünde, kullanıcı bilgisayarından bir FASTQ dosyası seçerek başlar. Uygulama, File nesnesiyle rastgele bir bayt konumundan başlayarak küçük bir veri yığınını okur (FileReader API'sini kullanarak). Bu veri yığınında, temel dize işlemlerini gerçekleştirmek ve ilgili ölçümleri hesaplamak için JavaScript kullanıyoruz. Böyle bir ölçüm, bir DNA parçası boyunca her bir pozisyonda tipik olarak kaç tane A, C, G ve T gördüğümüzü izlememize yardımcı olur.
Bu veri yığını için metrikler hesaplandıktan sonra, sonuçları Plotly.js ile etkileşimli olarak çizer ve dosyadaki bir sonraki parçaya geçeriz. Dosyayı küçük parçalar halinde işlemenin nedeni basitçe kullanıcı deneyimini geliştirmektir: FASTQ dosyaları genellikle yüzlerce gigabayt boyutunda olduğundan, tüm dosyayı bir kerede işlemek çok uzun sürer. 0,5 MB ile 1 MB arasında bir yığın boyutunun uygulamayı daha sorunsuz hale getireceğini ve bilgileri kullanıcıya daha hızlı döndüreceğini bulduk, ancak bu sayı uygulamanızın ayrıntılarına ve hesaplamaların ne kadar ağır olduğuna bağlı olarak değişecektir.
Orijinal JavaScript uygulamamızın mimarisi oldukça basitti:
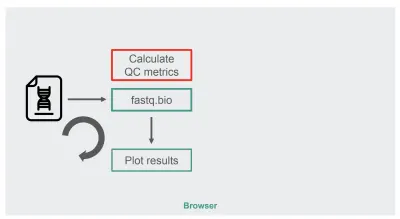
Kırmızı kutu, metrikleri oluşturmak için dize işlemlerini yaptığımız yerdir. Bu kutu, uygulamanın daha yoğun işlem gerektiren kısmıdır ve bu da onu WebAssembly ile çalışma zamanı optimizasyonu için doğal olarak iyi bir aday haline getirir.
fastq.bio: WebAssembly Uygulaması
Web uygulamamızı hızlandırmak için WebAssembly'den yararlanıp yararlanamayacağımızı keşfetmek için FASTQ dosyalarında QC ölçümlerini hesaplayan, kullanıma hazır bir araç aradık. Spesifik olarak, WebAssembly'ye taşımaya uygun olması için C/C++/Rust ile yazılmış ve bilimsel topluluk tarafından zaten onaylanmış ve güvenilen bir araç aradık.
Biraz araştırmadan sonra, sıralama verilerinin kalitesini değerlendirmemize yardımcı olabilecek (ve daha genel olarak bu veri dosyalarını işlemek için kullanılan) C dilinde yazılmış, yaygın olarak kullanılan, açık kaynaklı bir araç olan seqtk'yi kullanmaya karar verdik.
WebAssembly'yi derlemeden önce, komut satırında çalıştırmak için normalde seqtk'yi ikiliye nasıl derleyeceğimizi düşünelim. Makefile'a göre, ihtiyacınız olan gcc sihiri budur:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lzÖte yandan, seqtk'yi WebAssembly'ye derlemek için, WebAssembly'de çalışmayı kolaylaştırmak için mevcut derleme araçları için açılan değiştirmeler sağlayan Emscripten araç zincirini kullanabiliriz. Emscripten kurulu değilse, Dockerhub'da hazırladığımız ve ihtiyacınız olan araçlara sahip bir docker görüntüsünü indirebilirsiniz (sıfırdan da yükleyebilirsiniz, ancak bu genellikle biraz zaman alır):

$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26 Konteynerin içinde, gcc yerine emcc derleyicisini kullanabiliriz:
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1Gördüğünüz gibi, ikili ve WebAssembly için derleme arasındaki farklar minimumdur:
- Çıktının ikili dosya
seqtkolması yerine, Emscripten'den WebAssembly modülümüzün somutlaştırılmasını işleyen bir.wasmve bir.jsoluşturmasını istiyoruz. - zlib kitaplığını desteklemek için
USE_ZLIBbayrağını kullanıyoruz; zlib o kadar yaygın ki WebAssembly'ye taşınmış durumda ve Emscripten onu bizim için projemize dahil edecek - Emscripten'in POSIX benzeri bir dosya sistemi olan sanal dosya sistemini etkinleştiririz (burada kaynak kodu), ancak tarayıcıda RAM'de çalışır ve sayfayı yenilediğinizde kaybolur (durumunu IndexedDB kullanarak tarayıcıya kaydetmediğiniz sürece, ancak bu kadar) başka bir makale için).
Neden sanal dosya sistemi? Bunu yanıtlamak için, derlenmiş WebAssembly modülünü çağırmak için JavaScript kullanarak komut satırında seqtk'yi nasıl çağıracağımızı karşılaştıralım:
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"]) Bir sanal dosya sistemine erişim güçlüdür çünkü bu, dosya yolları yerine dize girişlerini işlemek için seqtk'yi yeniden yazmamız gerekmediği anlamına gelir. Bir veri yığınını sanal dosya sistemine data.fastq dosyası olarak bağlayabilir ve bunun üzerinde seqtk'nin main() işlevini çağırabiliriz.
WebAssembly'de derlenen seqtk ile, işte yeni fastq.bio mimarisi:
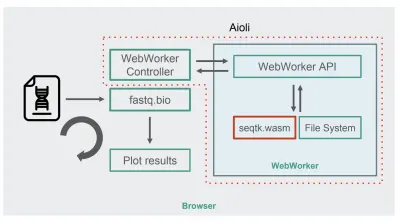
Diyagramda gösterildiği gibi, hesaplamaları tarayıcının ana iş parçacığında çalıştırmak yerine, hesaplamalarımızı bir arka plan iş parçacığında çalıştırmamıza izin veren ve tarayıcının yanıt verebilirliğini olumsuz yönde etkilemeyen WebWorker'ları kullanıyoruz. Spesifik olarak, WebWorker denetleyicisi Worker'ı başlatır ve ana iş parçacığı ile iletişimi yönetir. Çalışan tarafında, bir API aldığı istekleri yürütür.
Ardından Worker'dan yeni eklediğimiz dosya üzerinde bir seqtk komutu çalıştırmasını isteyebiliriz. seqtk çalışmayı bitirdiğinde, Çalışan sonucu bir Söz yoluyla ana iş parçacığına geri gönderir. Mesajı aldığında, ana iş parçacığı grafikleri güncellemek için elde edilen çıktıyı kullanır. JavaScript sürümüne benzer şekilde, dosyaları parçalar halinde işler ve her yinelemede görselleştirmeleri güncelleriz.
Verim iyileştirmesi
WebAssembly kullanmanın herhangi bir işe yarayıp yaramadığını değerlendirmek için, saniyede kaç okuma işleyebileceğimize ilişkin metriği kullanarak JavaScript ve WebAssembly uygulamalarını karşılaştırırız. Her iki uygulama da bu amaç için JavaScript kullandığından, etkileşimli grafikler oluşturmak için gereken süreyi göz ardı ediyoruz.
Kutunun dışında, zaten ~ 9X hızlanma görüyoruz:
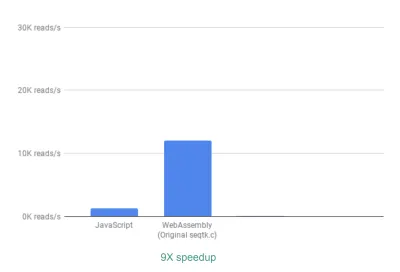
Bunu başarmanın nispeten kolay olduğu göz önüne alındığında, bu zaten çok iyidir (bu, WebAssembly'yi bir kez anladığınızda!).
Daha sonra, seqtk'nin çok sayıda genel olarak yararlı QC metrikleri vermesine rağmen, bu metriklerin çoğunun uygulamamız tarafından gerçekten kullanılmadığını veya grafiklendirilmediğini fark ettik. İhtiyacımız olmayan metrikler için çıktıların bir kısmını kaldırarak, 13X'lik daha da büyük bir hızlanma görebildik:
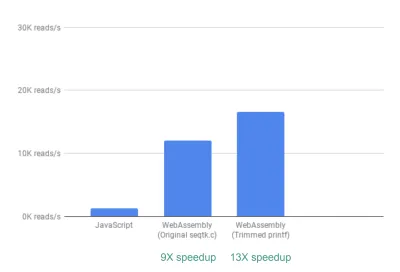
Bu, ihtiyaç duyulmayan printf ifadelerini kelimenin tam anlamıyla yorumlayarak elde etmenin ne kadar kolay olduğu düşünüldüğünde, yine büyük bir gelişmedir.
Son olarak, incelediğimiz bir gelişme daha var. Şimdiye kadar, fastq.bio'nun ilgilenilen metrikleri elde etme yolu, her biri farklı bir metrik kümesi hesaplayan iki farklı C fonksiyonunu çağırmaktır. Spesifik olarak, bir işlev bilgileri bir histogram (yani aralıklara bindirdiğimiz bir değerler listesi) biçiminde döndürürken, diğer işlev bilgiyi DNA dizi konumunun bir işlevi olarak döndürür. Ne yazık ki bu, aynı dosya parçasının iki kez okunduğu anlamına gelir ve bu gereksizdir.
Bu yüzden, iki işlevin kodunu - dağınık da olsa - tek bir işlevde birleştirdik (C'mi tazelemek zorunda kalmadan bile!). İki çıktıda farklı sayıda sütun olduğundan, ikisini ayırmak için JavaScript tarafında biraz çekişme yaptık. Ama buna değdi: bunu yapmak >20X hızlanma elde etmemizi sağladı!
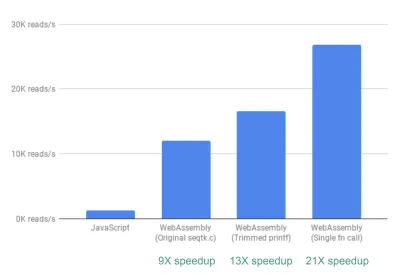
Uyarı kelimesi
Şimdi bir uyarı için iyi bir zaman olurdu. WebAssembly kullandığınızda her zaman 20 kat hızlanma elde etmeyi beklemeyin. Yalnızca 2 kat hızlanma veya %20 hızlanma elde edebilirsiniz. Veya belleğe çok büyük dosyalar yüklerseniz veya WebAssembly ile JavaScript arasında çok fazla iletişim gerektirirseniz yavaşlayabilir.
Çözüm
Kısacası, yavaş JavaScript hesaplamalarını derlenmiş WebAssembly çağrılarıyla değiştirmenin önemli hızlanmalara yol açabileceğini gördük. Bu hesaplamalar için gereken kod zaten C'de mevcut olduğundan, güvenilir bir aracı yeniden kullanmanın ek avantajını elde ettik. Ayrıca değindiğimiz gibi, WebAssembly bu iş için her zaman doğru araç olmayacaktır ( gasp! ), bu yüzden onu akıllıca kullanın.
Daha fazla okuma
- “WebAssembly ile Seviye Atlayın,” Robert Aboukhalil
WebAssembly uygulamaları oluşturmaya yönelik pratik bir kılavuz. - Aioli (GitHub'da)
Hızlı genomik web araçları oluşturmak için bir çerçeve. - fastq.bio kaynak kodu (GitHub'da)
DNA dizileme verilerinin kalite kontrolü için etkileşimli bir web aracı. - “WebAssembly'ye Kısaltılmış Bir Karikatür Girişi,” Lin Clark
