วิธีที่เราใช้ WebAssembly เพื่อเพิ่มความเร็วเว็บแอปของเราได้ถึง 20 เท่า (กรณีศึกษา)
เผยแพร่แล้ว: 2022-03-10หากคุณไม่เคยได้ยินมาก่อน นี่คือ TL;DR: WebAssembly เป็นภาษาใหม่ที่ทำงานในเบราว์เซอร์ควบคู่ไปกับ JavaScript ถูกต้องเลย. JavaScript ไม่ใช่ภาษาเดียวที่ทำงานในเบราว์เซอร์อีกต่อไป!
แต่นอกเหนือจากการเป็น “ไม่ใช่ JavaScript” แล้ว ปัจจัยที่แตกต่างคือคุณสามารถคอมไพล์โค้ดจากภาษาต่างๆ เช่น C/C++/Rust ( และอื่นๆ อีกมากมาย! ) ไปยัง WebAssembly และเรียกใช้ในเบราว์เซอร์ เนื่องจาก WebAssembly เป็นประเภทสแตติก ใช้หน่วยความจำเชิงเส้น และจัดเก็บในรูปแบบไบนารีขนาดกะทัดรัด จึงรวดเร็วมาก และในที่สุดอาจทำให้เราเรียกใช้โค้ดที่ความเร็ว "ใกล้เคียง" ได้ เช่น ที่ความเร็วใกล้เคียงกับที่คุณ d รับโดยการรันไบนารีบนบรรทัดคำสั่ง ความสามารถในการใช้ประโยชน์จากเครื่องมือและไลบรารีที่มีอยู่เพื่อใช้ในเบราว์เซอร์และศักยภาพที่เกี่ยวข้องในการเร่งความเร็ว เป็นเหตุผลสองประการที่ทำให้ WebAssembly น่าสนใจสำหรับเว็บ
จนถึงตอนนี้ WebAssembly ถูกใช้สำหรับแอปพลิเคชันทุกประเภท ตั้งแต่การเล่นเกม (เช่น Doom 3) ไปจนถึงการพอร์ตแอปพลิเคชันเดสก์ท็อปไปยังเว็บ (เช่น Autocad และ Figma) มีการใช้แม้นอกเบราว์เซอร์ เช่น เป็นภาษาที่มีประสิทธิภาพและยืดหยุ่นสำหรับการประมวลผลแบบไร้เซิร์ฟเวอร์
บทความนี้เป็นกรณีศึกษาเกี่ยวกับการใช้ WebAssembly เพื่อเพิ่มความเร็วให้กับเครื่องมือเว็บการวิเคราะห์ข้อมูล ด้วยเหตุนี้ เราจะใช้เครื่องมือที่มีอยู่ซึ่งเขียนด้วยภาษา C ซึ่งดำเนินการคำนวณแบบเดียวกัน คอมไพล์ไปยัง WebAssembly และใช้เพื่อแทนที่การคำนวณ JavaScript ที่ช้า
หมายเหตุ : บทความนี้จะเจาะลึกในหัวข้อขั้นสูง เช่น การคอมไพล์โค้ด C แต่อย่ากังวลถ้าคุณไม่มีประสบการณ์กับมัน คุณจะยังสามารถติดตามและทำความเข้าใจกับสิ่งที่เป็นไปได้ด้วย WebAssembly
พื้นหลัง
เว็บแอปที่เราจะใช้งานคือ fastq.bio ซึ่งเป็นเครื่องมือเว็บเชิงโต้ตอบที่ให้นักวิทยาศาสตร์สามารถดูตัวอย่างคุณภาพของข้อมูลการจัดลำดับดีเอ็นเอได้อย่างรวดเร็ว การจัดลำดับเป็นกระบวนการที่เราอ่าน "จดหมาย" (เช่น นิวคลีโอไทด์) ในตัวอย่างดีเอ็นเอ
นี่คือภาพหน้าจอของแอปพลิเคชันที่ใช้งานจริง:
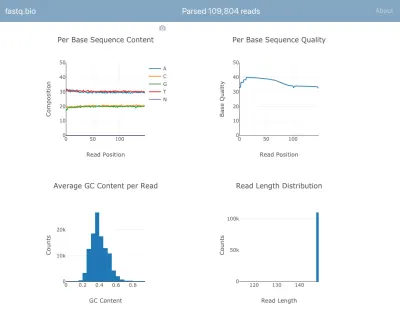
เราจะไม่พูดถึงรายละเอียดของการคำนวณ แต่โดยสรุป แผนภาพด้านบนช่วยให้นักวิทยาศาสตร์เข้าใจได้ว่าการจัดลำดับดำเนินไปได้ดีเพียงใด และใช้เพื่อระบุปัญหาด้านคุณภาพของข้อมูลได้อย่างรวดเร็ว
แม้ว่าจะมีเครื่องมือบรรทัดคำสั่งมากมายที่สร้างรายงานการควบคุมคุณภาพดังกล่าว เป้าหมายของ fastq.bio คือการให้การแสดงตัวอย่างแบบโต้ตอบของคุณภาพข้อมูลโดยไม่ต้องออกจากเบราว์เซอร์ สิ่งนี้มีประโยชน์อย่างยิ่งสำหรับนักวิทยาศาสตร์ที่ไม่สะดวกกับบรรทัดคำสั่ง
การป้อนข้อมูลในแอปเป็นไฟล์ข้อความธรรมดาที่ส่งออกโดยเครื่องมือจัดลำดับและมีรายการลำดับดีเอ็นเอและคะแนนคุณภาพสำหรับนิวคลีโอไทด์แต่ละตัวในลำดับดีเอ็นเอ รูปแบบของไฟล์นั้นเรียกว่า “FASTQ” ดังนั้นชื่อ fastq.bio
หากคุณอยากรู้เกี่ยวกับรูปแบบ FASTQ (ไม่จำเป็นต้องเข้าใจบทความนี้) ให้ตรวจสอบหน้า Wikipedia สำหรับ FASTQ (คำเตือน: รูปแบบไฟล์ FASTQ เป็นที่ทราบกันดีอยู่แล้วในฟิลด์เพื่อกระตุ้นให้เกิดการเอามือปิดหน้า)
fastq.bio: การใช้งาน JavaScript
ใน fastq.bio เวอร์ชันดั้งเดิม ผู้ใช้เริ่มต้นด้วยการเลือกไฟล์ FASTQ จากคอมพิวเตอร์ของตน ด้วยอ็อบเจ็กต์ File แอปจะอ่านข้อมูลกลุ่มเล็กๆ โดยเริ่มต้นที่ตำแหน่งไบต์สุ่ม (โดยใช้ FileReader API) ในกลุ่มข้อมูลนั้น เราใช้ JavaScript เพื่อดำเนินการจัดการสตริงพื้นฐานและคำนวณเมตริกที่เกี่ยวข้อง ตัววัดดังกล่าวตัวหนึ่งช่วยให้เราติดตามจำนวน A, C, G และ T ที่เรามักจะเห็นในแต่ละตำแหน่งตามส่วนย่อยของ DNA
เมื่อคำนวณเมตริกสำหรับกลุ่มข้อมูลนั้นแล้ว เราจะพล็อตผลลัพธ์แบบโต้ตอบด้วย Plotly.js และไปยังกลุ่มถัดไปในไฟล์ เหตุผลในการประมวลผลไฟล์เป็นชิ้นเล็กๆ ก็เพื่อปรับปรุงประสบการณ์ของผู้ใช้: การประมวลผลไฟล์ทั้งหมดในครั้งเดียวอาจใช้เวลานานเกินไป เนื่องจากไฟล์ FASTQ โดยทั่วไปจะมีหน่วยเป็นร้อยกิกะไบต์ เราพบว่าขนาดก้อนระหว่าง 0.5 MB ถึง 1 MB จะทำให้แอปพลิเคชันทำงานได้อย่างราบรื่นยิ่งขึ้น และจะส่งข้อมูลกลับไปยังผู้ใช้เร็วขึ้น แต่ตัวเลขนี้จะแตกต่างกันไปขึ้นอยู่กับรายละเอียดของแอปพลิเคชันของคุณและปริมาณการคำนวณ
สถาปัตยกรรมของการใช้งาน JavaScript ดั้งเดิมของเราค่อนข้างตรงไปตรงมา:
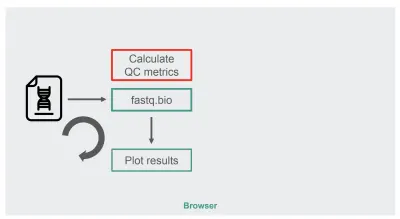
กล่องสีแดงเป็นที่ที่เราทำการปรับแต่งสตริงเพื่อสร้างตัววัด กล่องนั้นเป็นส่วนที่เน้นการประมวลผลมากกว่าของแอปพลิเคชัน ซึ่งทำให้เป็นตัวเลือกที่ดีสำหรับการเพิ่มประสิทธิภาพรันไทม์ด้วย WebAssembly
fastq.bio: The WebAssembly Implementation
ในการสำรวจว่าเราสามารถใช้ประโยชน์จาก WebAssembly เพื่อเพิ่มความเร็วเว็บแอปของเราได้หรือไม่ เราค้นหาเครื่องมือที่พร้อมใช้งานซึ่งคำนวณเมตริก QC ในไฟล์ FASTQ โดยเฉพาะอย่างยิ่ง เราค้นหาเครื่องมือที่เขียนด้วยภาษา C/C++/Rust เพื่อให้สามารถย้ายไปยัง WebAssembly และเครื่องมือที่ได้รับการตรวจสอบและเชื่อถือโดยชุมชนวิทยาศาสตร์แล้ว
หลังจากการค้นคว้า เราตัดสินใจที่จะใช้ seqtk ซึ่งเป็นเครื่องมือโอเพนซอร์สที่ใช้กันทั่วไปซึ่งเขียนด้วยภาษา C ที่สามารถช่วยเราประเมินคุณภาพของการจัดลำดับข้อมูล (และโดยทั่วไปมักใช้เพื่อจัดการไฟล์ข้อมูลเหล่านั้น)
ก่อนที่เราจะคอมไพล์ไปที่ WebAssembly เรามาพิจารณาก่อนว่าเราจะคอมไพล์ seqtk เป็นไบนารี่เพื่อรันบนบรรทัดคำสั่งได้อย่างไร ตาม Makefile นี่คือคาถา gcc ที่คุณต้องการ:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lzในทางกลับกัน ในการคอมไพล์ seqtk เป็น WebAssembly เราสามารถใช้ Emscripten toolchain ซึ่งให้การแทนที่แบบดรอปอินสำหรับเครื่องมือบิลด์ที่มีอยู่เพื่อทำให้การทำงานใน WebAssembly ง่ายขึ้น หากคุณไม่ได้ติดตั้ง Emscripten คุณสามารถดาวน์โหลดอิมเมจ Docker ที่เราเตรียมไว้บน Dockerhub ที่มีเครื่องมือต่างๆ ที่คุณต้องการ (คุณสามารถติดตั้งตั้งแต่เริ่มต้น แต่โดยปกติแล้วจะใช้เวลาสักครู่):

$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26 ภายในคอนเทนเนอร์ เราสามารถใช้คอมไพเลอร์ emcc แทน gcc :
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1อย่างที่คุณเห็น ความแตกต่างระหว่างการคอมไพล์เป็นไบนารีและ WebAssembly มีน้อยมาก:
- แทนที่จะให้ผลลัพธ์เป็นไฟล์ไบนารี
seqtkเราขอให้ Emscripten สร้าง.wasmและ ..jsที่จัดการการสร้างอินสแตนซ์ของโมดูล WebAssembly ของเรา - เพื่อรองรับไลบรารี zlib เราใช้แฟ
USE_ZLIB; zlib เป็นเรื่องธรรมดามากที่มีการย้ายไปยัง WebAssembly แล้ว และ Emscripten จะรวมไว้ในโครงการของเรา - เราเปิดใช้งานระบบไฟล์เสมือนของ Emscripten ซึ่งเป็นระบบไฟล์ที่เหมือน POSIX (ซอร์สโค้ดที่นี่) ยกเว้นว่ามันทำงานใน RAM ภายในเบราว์เซอร์และหายไปเมื่อคุณรีเฟรชหน้า (เว้นแต่คุณจะบันทึกสถานะในเบราว์เซอร์โดยใช้ IndexedDB แต่นั่นคือ สำหรับบทความอื่น)
ทำไมระบบไฟล์เสมือน? เพื่อตอบคำถามนั้น ให้ลองเปรียบเทียบวิธีที่เราจะเรียก seqtk บนบรรทัดคำสั่งกับการใช้ JavaScript เพื่อเรียกโมดูล WebAssembly ที่คอมไพล์แล้ว:
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"]) การเข้าถึงระบบไฟล์เสมือนนั้นมีประสิทธิภาพเพราะหมายความว่าเราไม่ต้องเขียน seqtk ใหม่เพื่อจัดการอินพุตสตริงแทนพาธของไฟล์ เราสามารถติดตั้งกลุ่มข้อมูลเป็นไฟล์ data.fastq บนระบบไฟล์เสมือนและเรียกใช้ฟังก์ชัน main() ของ seqtk ได้
ด้วย seqtk ที่คอมไพล์ไปยัง WebAssembly นี่คือสถาปัตยกรรม fastq.bio ใหม่:
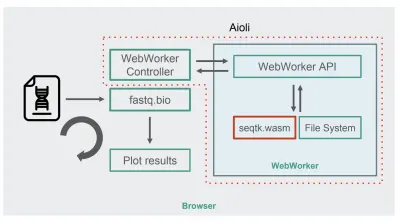
ตามที่แสดงในแผนภาพ แทนที่จะเรียกใช้การคำนวณในเธรดหลักของเบราว์เซอร์ เราใช้ WebWorkers ซึ่งช่วยให้เราเรียกใช้การคำนวณในเธรดพื้นหลัง และหลีกเลี่ยงผลเสียต่อการตอบสนองของเบราว์เซอร์ โดยเฉพาะตัวควบคุม WebWorker จะเปิดใช้ Worker และจัดการการสื่อสารกับเธรดหลัก ในด้านของผู้ปฏิบัติงาน API จะดำเนินการตามคำขอที่ได้รับ
จากนั้นเราสามารถขอให้ผู้ปฏิบัติงานเรียกใช้คำสั่ง seqtk ในไฟล์ที่เราเพิ่งเมาต์ เมื่อ seqtk ทำงานเสร็จ ผู้ปฏิบัติงานจะส่งผลลัพธ์กลับไปยังเธรดหลักผ่าน Promise เมื่อได้รับข้อความแล้ว เธรดหลักจะใช้ผลลัพธ์ที่ได้เพื่ออัปเดตแผนภูมิ เช่นเดียวกับเวอร์ชัน JavaScript เราประมวลผลไฟล์เป็นชิ้นๆ และอัปเดตการแสดงภาพในการทำซ้ำแต่ละครั้ง
การเพิ่มประสิทธิภาพการทำงาน
ในการประเมินว่าการใช้ WebAssembly ส่งผลดีหรือไม่ เราเปรียบเทียบการใช้งาน JavaScript และ WebAssembly โดยใช้เมตริกของจำนวนการอ่านที่เราสามารถประมวลผลต่อวินาที เราเพิกเฉยต่อเวลาที่ใช้ในการสร้างกราฟเชิงโต้ตอบ เนื่องจากการใช้งานทั้งสองแบบใช้ JavaScript เพื่อจุดประสงค์นั้น
นอกกรอบ เราเห็นการเร่งความเร็วประมาณ 9 เท่า:
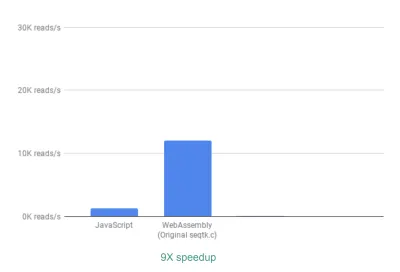
ซึ่งถือว่าดีมากอยู่แล้ว เนื่องจากทำได้ค่อนข้างง่าย (นั่นคือเมื่อคุณเข้าใจ WebAssembly แล้ว!)
ต่อไป เราสังเกตเห็นว่าแม้ว่า seqtk จะแสดงผลตัววัด QC ที่มีประโยชน์โดยทั่วไปจำนวนมาก แต่ตัววัดเหล่านี้จำนวนมากไม่ได้ใช้งานจริงหรือสร้างกราฟโดยแอปของเรา การลบเอาต์พุตบางส่วนสำหรับเมตริกที่เราไม่ต้องการออก ทำให้เราได้เห็นการเร่งความเร็วที่มากขึ้นถึง 13 เท่า:
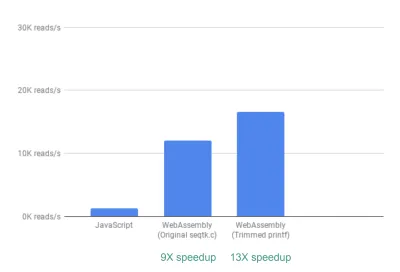
นี่เป็นอีกการปรับปรุงที่ยอดเยี่ยมเมื่อพิจารณาจากความง่ายในการบรรลุ—โดยการแสดงความคิดเห็นตามตัวอักษรของคำสั่ง printf ที่ไม่ต้องการ
ในที่สุดก็มีการปรับปรุงอีกประการหนึ่งที่เราพิจารณา จนถึงตอนนี้ วิธีที่ fastq.bio ได้รับเมตริกที่น่าสนใจคือการเรียกใช้ฟังก์ชัน C ที่แตกต่างกัน 2 ฟังก์ชัน โดยแต่ละฟังก์ชันจะคำนวณชุดเมตริกที่ต่างกัน โดยเฉพาะอย่างยิ่ง ฟังก์ชันหนึ่งส่งคืนข้อมูลในรูปแบบของฮิสโตแกรม (เช่น รายการค่าที่เรารวมเป็นช่วง) ในขณะที่ฟังก์ชันอื่นส่งคืนข้อมูลเป็นฟังก์ชันของตำแหน่งลำดับดีเอ็นเอ น่าเสียดาย นี่หมายความว่าไฟล์กลุ่มเดียวกันถูกอ่านสองครั้ง ซึ่งไม่จำเป็น
ดังนั้นเราจึงรวมโค้ดสำหรับสองฟังก์ชันเข้าเป็นหนึ่งเดียว—แม้ว่าจะยุ่ง—ฟังก์ชัน (โดยที่ไม่ต้องปัดฝุ่น C ของฉันด้วยซ้ำ!) เนื่องจากเอาต์พุตทั้งสองมีจำนวนคอลัมน์ต่างกัน เราจึงทำการโต้เถียงกันที่ฝั่ง JavaScript เพื่อแยกส่วนทั้งสองออก แต่มันก็คุ้มค่า: การทำเช่นนี้ทำให้เราได้รับความเร็ว >20X!
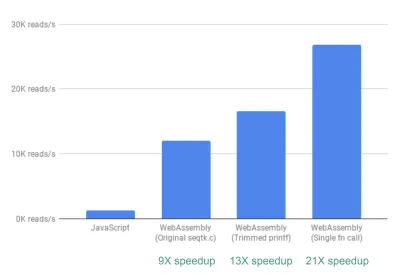
คำเตือน
ตอนนี้จะเป็นช่วงเวลาที่ดีสำหรับการเตือน อย่าคาดหวังว่าจะได้รับความเร็ว 20X เสมอเมื่อคุณใช้ WebAssembly คุณอาจได้รับความเร็วเพิ่มขึ้น 2 เท่าหรือเพิ่มความเร็ว 20% เท่านั้น หรือคุณอาจช้าลงหากคุณโหลดไฟล์ขนาดใหญ่มากในหน่วยความจำ หรือต้องการการสื่อสารจำนวนมากระหว่าง WebAssembly และ JavaScript
บทสรุป
กล่าวโดยสรุป เราพบว่าการแทนที่การคำนวณ JavaScript ที่ช้าด้วยการเรียกไปยัง WebAssembly ที่คอมไพล์แล้วสามารถนำไปสู่การเร่งความเร็วได้อย่างมาก เนื่องจากโค้ดที่จำเป็นสำหรับการคำนวณนั้นมีอยู่แล้วใน C เราจึงได้รับประโยชน์เพิ่มเติมจากการนำเครื่องมือที่เชื่อถือได้มาใช้ซ้ำ ตามที่เราได้กล่าวไปแล้ว WebAssembly ไม่ได้เป็นเครื่องมือที่เหมาะสมสำหรับงานนี้เสมอไป ( หอบ! ) ดังนั้นจงใช้มันอย่างชาญฉลาด
อ่านเพิ่มเติม
- “ยกระดับด้วย WebAssembly” โรเบิร์ต อาบูคาลิล
คู่มือปฏิบัติในการสร้างแอปพลิเคชัน WebAssembly - Aioli (บน GitHub)
กรอบงานสำหรับสร้างเครื่องมือเว็บจีโนมที่รวดเร็ว - ซอร์สโค้ด fastq.bio (บน GitHub)
เครื่องมือเว็บเชิงโต้ตอบสำหรับการควบคุมคุณภาพของข้อมูลการจัดลำดับดีเอ็นเอ - “การ์ตูนโดยย่อ Introduction To WebAssembly,” Lin Clark
