Cum am folosit WebAssembly pentru a accelera aplicația noastră web de 20 ori (studiu de caz)
Publicat: 2022-03-10Dacă nu ați auzit, iată TL;DR: WebAssembly este un nou limbaj care rulează în browser alături de JavaScript. Da, așa e. JavaScript nu mai este singura limbă care rulează în browser!
Dar, dincolo de a fi „nu JavaScript”, factorul său distinctiv este că puteți compila cod din limbaje precum C/C++/Rust ( și multe altele! ) în WebAssembly și le puteți rula în browser. Deoarece WebAssembly este scris static, folosește o memorie liniară și este stocat într-un format binar compact, este, de asemenea, foarte rapid și, în cele din urmă, ne-ar putea permite să rulăm cod la viteze „aproape native”, adică la viteze apropiate de ceea ce dvs. d obține prin rularea binarului pe linia de comandă. Capacitatea de a folosi instrumentele și bibliotecile existente pentru utilizare în browser și potențialul asociat de accelerare sunt două motive care fac ca WebAssembly să fie atât de convingător pentru web.
Până acum, WebAssembly a fost folosit pentru tot felul de aplicații, de la jocuri (de exemplu, Doom 3), până la portarea aplicațiilor desktop pe web (ex. Autocad și Figma). Este folosit chiar și în afara browserului, de exemplu ca un limbaj eficient și flexibil pentru calculul fără server.
Acest articol este un studiu de caz despre utilizarea WebAssembly pentru a accelera un instrument web de analiză a datelor. În acest scop, vom lua un instrument existent scris în C care efectuează aceleași calcule, îl vom compila în WebAssembly și îl vom folosi pentru a înlocui calculele JavaScript lente.
Notă : Acest articol analizează câteva subiecte avansate, cum ar fi compilarea codului C, dar nu vă faceți griji dacă nu aveți experiență în acest sens; veți putea în continuare să urmăriți și să înțelegeți ce este posibil cu WebAssembly.
fundal
Aplicația web cu care vom lucra este fastq.bio, un instrument web interactiv care oferă oamenilor de știință o previzualizare rapidă a calității datelor lor de secvențiere ADN; secvențierea este procesul prin care citim „literele” (adică nucleotidele) dintr-o probă de ADN.
Iată o captură de ecran a aplicației în acțiune:
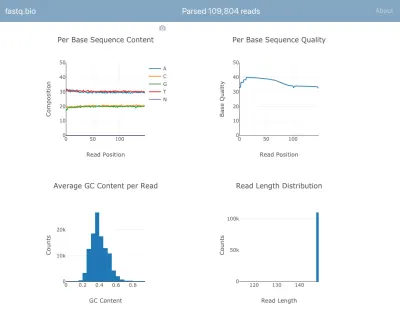
Nu vom intra în detaliile calculelor, dar pe scurt, diagramele de mai sus oferă oamenilor de știință cât de bine a mers secvențierea și sunt folosite pentru a identifica problemele de calitate a datelor dintr-o privire.
Deși există zeci de instrumente de linie de comandă disponibile pentru a genera astfel de rapoarte de control al calității, scopul fastq.bio este de a oferi o previzualizare interactivă a calității datelor fără a părăsi browserul. Acest lucru este util în special pentru oamenii de știință care nu sunt confortabili cu linia de comandă.
Intrarea în aplicație este un fișier text simplu care este scos de instrumentul de secvențiere și conține o listă de secvențe ADN și un scor de calitate pentru fiecare nucleotidă din secvențele ADN. Formatul acelui fișier este cunoscut ca „FASTQ”, de unde și numele fastq.bio.
Dacă sunteți curios despre formatul FASTQ (nu este necesar pentru a înțelege acest articol), consultați pagina Wikipedia pentru FASTQ. (Avertisment: Formatul de fișier FASTQ este cunoscut în domeniu pentru a induce palmele faciale.)
fastq.bio: Implementarea JavaScript
În versiunea originală a fastq.bio, utilizatorul începe prin a selecta un fișier FASTQ de pe computer. Cu obiectul File , aplicația citește o mică bucată de date începând de la o poziție aleatorie de octeți (folosind API-ul FileReader). În acea bucată de date, folosim JavaScript pentru a efectua manipulări de bază ale șirurilor și pentru a calcula valorile relevante. O astfel de măsurătoare ne ajută să urmărim câte A, C, G și T vedem de obicei la fiecare poziție de-a lungul unui fragment de ADN.
Odată ce valorile sunt calculate pentru acea bucată de date, graficăm rezultatele în mod interactiv cu Plotly.js și trecem la următoarea bucată din fișier. Motivul procesării fișierului în bucăți mici este pur și simplu pentru a îmbunătăți experiența utilizatorului: procesarea întregului fișier deodată ar dura prea mult, deoarece fișierele FASTQ sunt în general de sute de gigaocteți. Am descoperit că o dimensiune a fragmentului între 0,5 MB și 1 MB ar face aplicația mai fluidă și ar returna informațiile utilizatorului mai rapid, dar acest număr va varia în funcție de detaliile aplicației dvs. și de cât de grele sunt calculele.
Arhitectura implementării noastre originale JavaScript a fost destul de simplă:
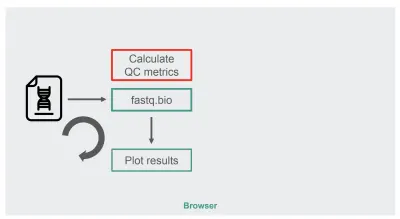
Caseta în roșu este locul în care facem manipulările șirurilor pentru a genera valorile. Acea casetă este partea mai intensă de calcul a aplicației, ceea ce a făcut-o în mod natural un bun candidat pentru optimizarea timpului de execuție cu WebAssembly.
fastq.bio: Implementarea WebAssembly
Pentru a explora dacă am putea folosi WebAssembly pentru a accelera aplicația noastră web, am căutat un instrument disponibil care calculează valorile QC pe fișierele FASTQ. Mai exact, am căutat un instrument scris în C/C++/Rust, astfel încât să poată fi portat în WebAssembly și unul care a fost deja validat și de încredere de comunitatea științifică.
După câteva cercetări, am decis să mergem cu seqtk, un instrument cu sursă deschisă folosit în mod obișnuit, scris în C, care ne poate ajuta să evaluăm calitatea secvențialării datelor (și este, în general, folosit pentru a manipula acele fișiere de date).
Înainte de a compila în WebAssembly, să ne gândim mai întâi cum am compila în mod normal seqtk în binar pentru a-l rula pe linia de comandă. Conform Makefile, aceasta este incantația gcc de care aveți nevoie:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lzPe de altă parte, pentru a compila seqtk în WebAssembly, putem folosi lanțul de instrumente Emscripten, care oferă înlocuiri introduse pentru instrumentele de construcție existente pentru a face lucrul în WebAssembly mai ușor. Dacă nu aveți instalat Emscripten, puteți descărca o imagine docker pe care am pregătit-o pe Dockerhub, care are instrumentele de care aveți nevoie (puteți să o instalați și de la zero, dar de obicei durează ceva timp):

$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26 În interiorul containerului, putem folosi compilatorul emcc ca înlocuitor pentru gcc :
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1După cum puteți vedea, diferențele dintre compilarea în binar și WebAssembly sunt minime:
- În loc ca rezultatul să fie fișierul binar
seqtk, îi cerem lui Emscripten să genereze un.wasmși un.jscare se ocupă de instanțierea modulului nostru WebAssembly - Pentru a susține biblioteca zlib, folosim flag
USE_ZLIB; zlib este atât de comun încât a fost deja portat în WebAssembly, iar Emscripten îl va include pentru noi în proiectul nostru - Activem sistemul de fișiere virtual Emscripten, care este un sistem de fișiere asemănător POSIX (codul sursă aici), cu excepția că rulează în RAM în browser și dispare când reîmprospătați pagina (cu excepția cazului în care îi salvați starea în browser folosind IndexedDB, dar asta este pentru alt articol).
De ce un sistem de fișiere virtual? Pentru a răspunde la asta, să comparăm modul în care am apela seqtk pe linia de comandă cu utilizarea JavaScript pentru a apela modulul WebAssembly compilat:
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"]) A avea acces la un sistem de fișiere virtual este puternic, deoarece înseamnă că nu trebuie să rescriem seqtk pentru a gestiona intrările de șir în loc de căile fișierelor. Putem monta o bucată de date ca fișier data.fastq pe sistemul de fișiere virtual și pur și simplu apelăm funcția main() a seqtk pe acesta.
Cu seqtk compilat în WebAssembly, iată noua arhitectură fastq.bio:
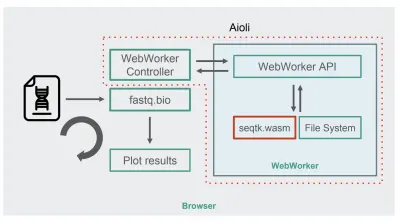
După cum se arată în diagramă, în loc să rulăm calculele în firul principal al browserului, folosim WebWorkers, care ne permit să rulăm calculele într-un thread de fundal și să evităm afectarea negativă a capacității de răspuns a browserului. Mai exact, controlerul WebWorker lansează Worker și gestionează comunicarea cu firul principal. Pe partea lucrătorului, un API execută solicitările pe care le primește.
Apoi îi putem cere lucrătorului să execute o comandă seqtk pe fișierul pe care tocmai l-am montat. Când seqtk termină de rulat, lucrătorul trimite rezultatul înapoi la firul principal printr-o Promisiune. Odată ce primește mesajul, firul principal folosește rezultatul rezultat pentru a actualiza diagramele. Similar cu versiunea JavaScript, procesăm fișierele în bucăți și actualizăm vizualizările la fiecare iterație.
Optimizarea performanței
Pentru a evalua dacă folosirea WebAssembly a avut un efect benefic, comparăm implementările JavaScript și WebAssembly folosind metrica câte citiri putem procesa pe secundă. Ignorăm timpul necesar pentru generarea graficelor interactive, deoarece ambele implementări folosesc JavaScript în acest scop.
Din cutie, vedem deja o accelerare de ~9X:
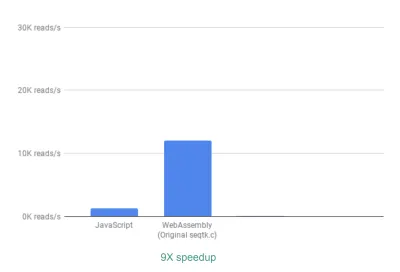
Acest lucru este deja foarte bun, având în vedere că a fost relativ ușor de realizat (adică odată ce înțelegi WebAssembly!).
Apoi, am observat că, deși seqtk produce o mulțime de valori QC utile în general, multe dintre aceste valori nu sunt de fapt utilizate sau reprezentate grafic de aplicația noastră. Eliminand o parte din ieșirea pentru valorile de care nu aveam nevoie, am putut vedea o accelerare și mai mare de 13X:
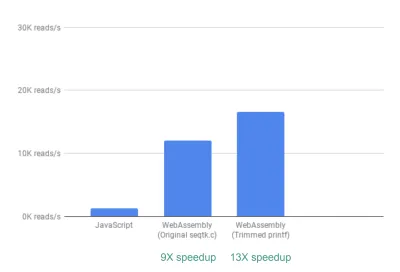
Aceasta, din nou, este o îmbunătățire extraordinară, având în vedere cât de ușor a fost de realizat, comentând literalmente declarații printf care nu erau necesare.
În cele din urmă, mai există o îmbunătățire la care ne-am uitat. Până acum, modul în care fastq.bio obține metricile de interes este apelând două funcții C diferite, fiecare calculând un set diferit de metrici. Mai exact, o funcție returnează informații sub forma unei histograme (adică o listă de valori pe care le împărțim în intervale), în timp ce cealaltă funcție returnează informații în funcție de poziția secvenței ADN. Din păcate, aceasta înseamnă că aceeași bucată de fișier este citită de două ori, ceea ce este inutil.
Așa că am îmbinat codul pentru cele două funcții într-o singură funcție – deși dezordonată – (fără a fi nevoie măcar să perfecționăm C-ul meu!). Deoarece cele două ieșiri au un număr diferit de coloane, am făcut câteva dispute pe partea JavaScript pentru a le dezlega pe cele două. Dar a meritat: acest lucru ne-a permis să obținem o accelerare >20X!
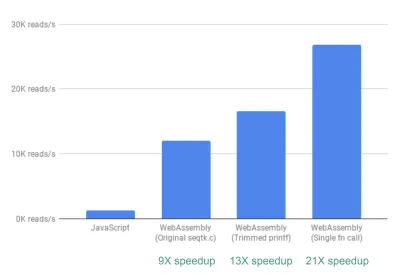
Un cuvânt de precauție
Acum ar fi un moment bun pentru o avertizare. Nu vă așteptați să obțineți întotdeauna o accelerare de 20 ori când utilizați WebAssembly. Este posibil să obțineți doar o accelerare de 2X sau o accelerare de 20%. Sau puteți obține o încetinire dacă încărcați fișiere foarte mari în memorie sau aveți nevoie de multă comunicare între WebAssembly și JavaScript.
Concluzie
Pe scurt, am văzut că înlocuirea calculelor lente JavaScript cu apeluri la WebAssembly compilat poate duce la accelerari semnificative. Deoarece codul necesar pentru acele calcule exista deja în C, am beneficiat de avantajul suplimentar de a reutiliza un instrument de încredere. După cum am atins, de asemenea, WebAssembly nu va fi întotdeauna instrumentul potrivit pentru această lucrare ( găfește! ), așa că folosește-l cu înțelepciune.
Lectură suplimentară
- „Măriți la nivel cu WebAssembly”, Robert Aboukhalil
Un ghid practic pentru construirea de aplicații WebAssembly. - Aioli (pe GitHub)
Un cadru pentru construirea de instrumente web rapide de genomică. - codul sursă fastq.bio (pe GitHub)
Un instrument web interactiv pentru controlul calității datelor de secvențiere ADN. - „O introducere prescurtată a desenului animat la WebAssembly”, Lin Clark
