Bagaimana Kami Menggunakan WebAssembly Untuk Mempercepat Aplikasi Web Kami Hingga 20X (Studi Kasus)
Diterbitkan: 2022-03-10Jika Anda belum pernah mendengarnya, inilah TL; DR: WebAssembly adalah bahasa baru yang berjalan di browser bersama JavaScript. Ya itu betul. JavaScript bukan lagi satu-satunya bahasa yang berjalan di browser!
Tetapi selain menjadi "bukan JavaScript", faktor pembedanya adalah Anda dapat mengkompilasi kode dari bahasa seperti C/C++/Rust ( dan banyak lagi! ) ke WebAssembly dan menjalankannya di browser. Karena WebAssembly diketik secara statis, menggunakan memori linier, dan disimpan dalam format biner yang ringkas, WebAssembly juga sangat cepat, dan pada akhirnya memungkinkan kita untuk menjalankan kode pada kecepatan "mendekati asli", yaitu pada kecepatan yang mendekati kecepatan Anda. d dapatkan dengan menjalankan biner pada baris perintah. Kemampuan untuk memanfaatkan alat dan pustaka yang ada untuk digunakan di browser dan potensi terkait untuk mempercepat, adalah dua alasan yang membuat WebAssembly begitu menarik untuk web.
Sejauh ini, WebAssembly telah digunakan untuk semua jenis aplikasi, mulai dari game (misalnya Doom 3), hingga porting aplikasi desktop ke web (misalnya Autocad dan Figma). Bahkan digunakan di luar browser, misalnya sebagai bahasa yang efisien dan fleksibel untuk komputasi tanpa server.
Artikel ini adalah studi kasus tentang penggunaan WebAssembly untuk mempercepat alat web analisis data. Untuk itu, kami akan menggunakan alat yang ada yang ditulis dalam C yang melakukan perhitungan yang sama, mengompilasinya ke WebAssembly, dan menggunakannya untuk menggantikan perhitungan JavaScript yang lambat.
Catatan : Artikel ini membahas beberapa topik lanjutan seperti kompilasi kode C, tetapi jangan khawatir jika Anda tidak memiliki pengalaman dengan itu; Anda masih dapat mengikuti dan memahami apa yang mungkin dilakukan dengan WebAssembly.
Latar belakang
Aplikasi web yang akan kami gunakan adalah fastq.bio, alat web interaktif yang memberi para ilmuwan pratinjau cepat tentang kualitas data pengurutan DNA mereka; sequencing adalah proses dimana kita membaca "huruf" (yaitu nukleotida) dalam sampel DNA.
Berikut tangkapan layar aplikasi yang sedang beraksi:
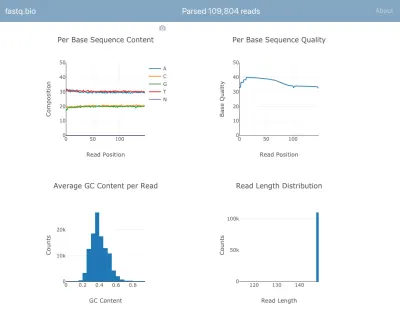
Kami tidak akan membahas detail perhitungannya, tetapi singkatnya, plot di atas memberi para ilmuwan pemahaman tentang seberapa baik pengurutan berjalan dan digunakan untuk mengidentifikasi masalah kualitas data secara sekilas.
Meskipun ada lusinan alat baris perintah yang tersedia untuk menghasilkan laporan kontrol kualitas seperti itu, tujuan fastq.bio adalah memberikan pratinjau interaktif kualitas data tanpa meninggalkan browser. Ini sangat berguna bagi para ilmuwan yang tidak nyaman dengan baris perintah.
Masukan ke aplikasi adalah file teks biasa yang dikeluarkan oleh instrumen sekuensing dan berisi daftar sekuens DNA dan skor kualitas untuk setiap nukleotida dalam sekuens DNA. Format file itu dikenal sebagai “FASTQ”, oleh karena itu diberi nama fastq.bio.
Jika Anda penasaran dengan format FASTQ (tidak perlu memahami artikel ini), lihat halaman Wikipedia untuk FASTQ. (Peringatan: Format file FASTQ dikenal di lapangan untuk menginduksi facepalms.)
fastq.bio: Implementasi JavaScript
Dalam versi asli fastq.bio, pengguna memulai dengan memilih file FASTQ dari komputer mereka. Dengan objek File , aplikasi membaca sebagian kecil data yang dimulai dari posisi byte acak (menggunakan FileReader API). Dalam potongan data tersebut, kami menggunakan JavaScript untuk melakukan manipulasi string dasar dan menghitung metrik yang relevan. Salah satu metrik semacam itu membantu kita melacak berapa banyak A, C, G, dan T yang biasanya kita lihat di setiap posisi di sepanjang fragmen DNA.
Setelah metrik dihitung untuk potongan data tersebut, kami memplot hasilnya secara interaktif dengan Plotly.js, dan melanjutkan ke potongan berikutnya dalam file. Alasan untuk memproses file dalam potongan kecil hanyalah untuk meningkatkan pengalaman pengguna: memproses seluruh file sekaligus akan memakan waktu terlalu lama, karena file FASTQ umumnya berukuran ratusan gigabyte. Kami menemukan bahwa ukuran potongan antara 0,5 MB dan 1 MB akan membuat aplikasi lebih mulus dan akan mengembalikan informasi kepada pengguna lebih cepat, tetapi jumlah ini akan bervariasi tergantung pada detail aplikasi Anda dan seberapa berat perhitungannya.
Arsitektur implementasi JavaScript asli kami cukup sederhana:
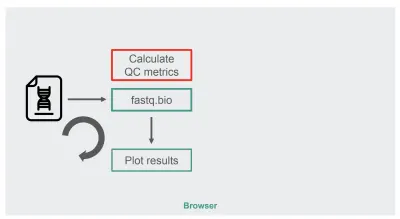
Kotak berwarna merah adalah tempat kita melakukan manipulasi string untuk menghasilkan metrik. Kotak itu adalah bagian aplikasi yang lebih intensif komputasi, yang secara alami menjadikannya kandidat yang baik untuk pengoptimalan runtime dengan WebAssembly.
fastq.bio: Implementasi WebAssembly
Untuk mengeksplorasi apakah kami dapat memanfaatkan WebAssembly untuk mempercepat aplikasi web kami, kami mencari alat siap pakai yang menghitung metrik QC pada file FASTQ. Secara khusus, kami mencari alat yang ditulis dalam C/C++/Rust sehingga dapat diporting ke WebAssembly, dan alat yang sudah divalidasi dan dipercaya oleh komunitas ilmiah.
Setelah beberapa penelitian, kami memutuskan untuk menggunakan seqtk, alat open-source yang umum digunakan yang ditulis dalam C yang dapat membantu kami mengevaluasi kualitas data pengurutan (dan lebih umum digunakan untuk memanipulasi file data tersebut).
Sebelum kita mengkompilasi ke WebAssembly, pertama-tama mari kita pertimbangkan bagaimana kita biasanya mengkompilasi seqtk ke biner untuk menjalankannya pada baris perintah. Menurut Makefile, ini adalah mantra gcc yang Anda butuhkan:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lzDi sisi lain, untuk mengkompilasi seqtk ke WebAssembly, kita dapat menggunakan toolchain Emscripten, yang menyediakan pengganti drop-in untuk alat build yang ada untuk mempermudah pekerjaan di WebAssembly. Jika Anda belum menginstal Emscripten, Anda dapat mengunduh gambar buruh pelabuhan yang kami siapkan di Dockerhub yang memiliki alat yang Anda perlukan (Anda juga dapat menginstalnya dari awal, tetapi biasanya memakan waktu cukup lama):

$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26 Di dalam container, kita dapat menggunakan compiler emcc sebagai pengganti gcc :
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1Seperti yang Anda lihat, perbedaan antara kompilasi ke biner dan WebAssembly minimal:
- Alih-alih output menjadi file biner
seqtk, kami meminta Emscripten untuk menghasilkan.wasmdan.jsyang menangani instantiasi modul WebAssembly kami - Untuk mendukung pustaka zlib, kami menggunakan flag
USE_ZLIB; zlib sangat umum sehingga sudah di-porting ke WebAssembly, dan Emscripten akan menyertakannya untuk kami dalam proyek kami - Kami mengaktifkan sistem file virtual Emscripten, yang merupakan sistem file mirip POSIX (kode sumber di sini), kecuali itu berjalan di RAM di dalam browser dan menghilang saat Anda menyegarkan halaman (kecuali jika Anda menyimpan statusnya di browser menggunakan IndexedDB, tapi itu untuk artikel lain).
Mengapa sistem file virtual? Untuk menjawabnya, mari kita bandingkan bagaimana kita akan memanggil seqtk pada baris perintah vs. menggunakan JavaScript untuk memanggil modul WebAssembly yang dikompilasi:
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"]) Memiliki akses ke sistem file virtual sangat kuat karena itu berarti kita tidak perlu menulis ulang seqtk untuk menangani input string alih-alih jalur file. Kita dapat memasang sepotong data sebagai file data.fastq pada sistem file virtual dan cukup memanggil fungsi main() seqtk di atasnya.
Dengan seqtk dikompilasi ke WebAssembly, inilah arsitektur fastq.bio baru:
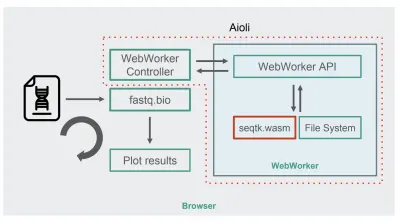
Seperti yang ditunjukkan dalam diagram, alih-alih menjalankan perhitungan di thread utama browser, kami menggunakan WebWorkers, yang memungkinkan kami untuk menjalankan perhitungan kami di thread latar belakang, dan menghindari pengaruh negatif terhadap respons browser. Secara khusus, pengontrol WebWorker meluncurkan Pekerja dan mengelola komunikasi dengan utas utama. Di sisi Pekerja, API mengeksekusi permintaan yang diterimanya.
Kami kemudian dapat meminta Pekerja untuk menjalankan perintah seqtk pada file yang baru saja kami pasang. Ketika seqtk selesai berjalan, Worker mengirimkan hasilnya kembali ke thread utama melalui Promise. Setelah menerima pesan, utas utama menggunakan output yang dihasilkan untuk memperbarui grafik. Mirip dengan versi JavaScript, kami memproses file dalam potongan dan memperbarui visualisasi di setiap iterasi.
Optimasi Kinerja
Untuk mengevaluasi apakah menggunakan WebAssembly ada gunanya, kami membandingkan implementasi JavaScript dan WebAssembly menggunakan metrik jumlah pembacaan yang dapat kami proses per detik. Kami mengabaikan waktu yang diperlukan untuk menghasilkan grafik interaktif, karena kedua implementasi menggunakan JavaScript untuk tujuan itu.
Di luar kotak, kita sudah melihat percepatan ~9X:
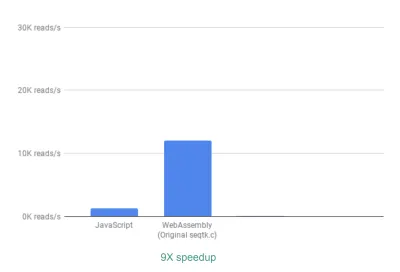
Ini sudah sangat bagus, mengingat itu relatif mudah dicapai (yaitu setelah Anda memahami WebAssembly!).
Selanjutnya, kami memperhatikan bahwa meskipun seqtk menghasilkan banyak metrik QC yang umumnya berguna, banyak dari metrik ini tidak benar-benar digunakan atau dibuat grafik oleh aplikasi kami. Dengan menghapus beberapa keluaran untuk metrik yang tidak kami perlukan, kami dapat melihat peningkatan yang lebih besar 13X:
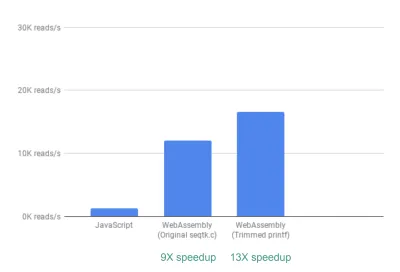
Sekali lagi ini adalah peningkatan besar mengingat betapa mudahnya mencapainya—dengan benar-benar mengomentari pernyataan printf yang tidak diperlukan.
Akhirnya, ada satu peningkatan lagi yang kami lihat. Sejauh ini, cara fastq.bio memperoleh metrik yang diinginkan adalah dengan memanggil dua fungsi C yang berbeda, yang masing-masing menghitung kumpulan metrik yang berbeda. Secara khusus, satu fungsi mengembalikan informasi dalam bentuk histogram (yaitu daftar nilai yang kita masukkan ke dalam rentang), sedangkan fungsi lainnya mengembalikan informasi sebagai fungsi posisi urutan DNA. Sayangnya, ini berarti potongan file yang sama dibaca dua kali, yang sebenarnya tidak perlu.
Jadi kami menggabungkan kode untuk dua fungsi menjadi satu—walaupun berantakan—fungsi (bahkan tanpa harus memoles C saya!). Karena kedua keluaran memiliki jumlah kolom yang berbeda, kami melakukan beberapa perdebatan di sisi JavaScript untuk menguraikan keduanya. Tapi itu sepadan: hal itu memungkinkan kami mencapai kecepatan >20X!
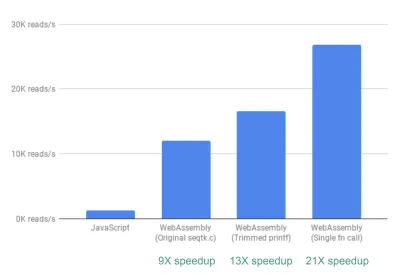
Sebuah Kata Perhatian
Sekarang akan menjadi saat yang tepat untuk peringatan. Jangan berharap untuk selalu mendapatkan kecepatan 20X saat Anda menggunakan WebAssembly. Anda mungkin hanya mendapatkan percepatan 2X atau percepatan 20%. Atau Anda mungkin melambat jika memuat file yang sangat besar di memori, atau memerlukan banyak komunikasi antara WebAssembly dan JavaScript.
Kesimpulan
Singkatnya, kami telah melihat bahwa mengganti komputasi JavaScript yang lambat dengan panggilan ke WebAssembly yang dikompilasi dapat menghasilkan percepatan yang signifikan. Karena kode yang diperlukan untuk perhitungan tersebut sudah ada di C, kami mendapat manfaat tambahan dengan menggunakan kembali alat tepercaya. Seperti yang juga telah kami singgung, WebAssembly tidak akan selalu menjadi alat yang tepat untuk pekerjaan itu ( terkesiap! ), jadi gunakan dengan bijak.
Bacaan lebih lanjut
- “Naik Level Dengan WebAssembly,” Robert Aboukhalil
Panduan praktis untuk membangun aplikasi WebAssembly. - Aioli (di GitHub)
Kerangka kerja untuk membangun alat web genomik cepat. - kode sumber fastq.bio (di GitHub)
Alat web interaktif untuk kontrol kualitas data sekuensing DNA. - “Sebuah Pengantar Singkat Kartun Untuk WebAssembly,” Lin Clark
