Wie wir WebAssembly verwendet haben, um unsere Webanwendung um das 20-fache zu beschleunigen (Fallstudie)
Veröffentlicht: 2022-03-10Falls Sie es noch nicht gehört haben, hier ist das TL;DR: WebAssembly ist eine neue Sprache, die neben JavaScript im Browser ausgeführt wird. Ja, das ist richtig. JavaScript ist nicht mehr die einzige Sprache, die im Browser läuft!
Aber abgesehen davon, dass es „kein JavaScript“ ist, zeichnet es sich dadurch aus, dass Sie Code aus Sprachen wie C/C++/Rust ( und mehr! ) in WebAssembly kompilieren und im Browser ausführen können. Da WebAssembly statisch typisiert ist, einen linearen Speicher verwendet und in einem kompakten Binärformat gespeichert wird, ist es auch sehr schnell und könnte es uns schließlich ermöglichen, Code mit „nahezu nativer“ Geschwindigkeit auszuführen, dh mit Geschwindigkeiten, die Ihrer eigenen Geschwindigkeit nahekommen. d erhalten, indem Sie die Binärdatei auf der Befehlszeile ausführen. Die Möglichkeit, vorhandene Tools und Bibliotheken für die Verwendung im Browser zu nutzen, und das damit verbundene Beschleunigungspotenzial sind zwei Gründe, die WebAssembly für das Web so überzeugend machen.
Bisher wurde WebAssembly für alle möglichen Anwendungen eingesetzt, von Spielen (z. B. Doom 3) bis hin zur Portierung von Desktop-Anwendungen ins Web (z. B. Autocad und Figma). Es wird sogar außerhalb des Browsers verwendet, beispielsweise als effiziente und flexible Sprache für Serverless Computing.
Dieser Artikel ist eine Fallstudie zur Verwendung von WebAssembly zur Beschleunigung eines Datenanalyse-Webtools. Zu diesem Zweck nehmen wir ein vorhandenes Tool, das in C geschrieben ist und dieselben Berechnungen durchführt, kompilieren es zu WebAssembly und verwenden es, um langsame JavaScript-Berechnungen zu ersetzen.
Hinweis : Dieser Artikel befasst sich mit einigen fortgeschrittenen Themen wie dem Kompilieren von C-Code, aber machen Sie sich keine Sorgen, wenn Sie damit keine Erfahrung haben; Sie können trotzdem mitverfolgen und ein Gefühl dafür bekommen, was mit WebAssembly möglich ist.
Hintergrund
Die Web-App, mit der wir arbeiten werden, ist fastq.bio, ein interaktives Web-Tool, das Wissenschaftlern eine schnelle Vorschau auf die Qualität ihrer DNA-Sequenzierungsdaten bietet; Sequenzierung ist der Prozess, bei dem wir die „Buchstaben“ (dh Nukleotide) in einer DNA-Probe lesen.
Hier ist ein Screenshot der Anwendung in Aktion:
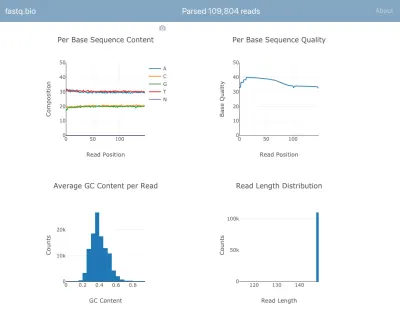
Wir werden nicht auf die Details der Berechnungen eingehen, aber kurz gesagt, die obigen Diagramme geben den Wissenschaftlern einen Eindruck davon, wie gut die Sequenzierung verlief, und werden verwendet, um Datenqualitätsprobleme auf einen Blick zu erkennen.
Obwohl es Dutzende von Befehlszeilentools gibt, um solche Qualitätskontrollberichte zu erstellen, ist das Ziel von fastq.bio, eine interaktive Vorschau der Datenqualität zu geben, ohne den Browser zu verlassen. Dies ist besonders nützlich für Wissenschaftler, die mit der Befehlszeile nicht vertraut sind.
Die Eingabe in die App ist eine Klartextdatei, die vom Sequenzierungsinstrument ausgegeben wird und eine Liste von DNA-Sequenzen und eine Qualitätsbewertung für jedes Nukleotid in den DNA-Sequenzen enthält. Das Format dieser Datei ist als „FASTQ“ bekannt, daher der Name fastq.bio.
Wenn Sie neugierig auf das FASTQ-Format sind (nicht erforderlich, um diesen Artikel zu verstehen), besuchen Sie die Wikipedia-Seite für FASTQ. (Warnung: Das FASTQ-Dateiformat ist in der Fachwelt dafür bekannt, Facepalms zu induzieren.)
fastq.bio: Die JavaScript-Implementierung
In der Originalversion von fastq.bio wählt der Benutzer zunächst eine FASTQ-Datei von seinem Computer aus. Mit dem File -Objekt liest die App einen kleinen Datenblock beginnend an einer zufälligen Byte-Position (unter Verwendung der FileReader-API). In diesem Datenblock verwenden wir JavaScript, um grundlegende String-Manipulationen durchzuführen und relevante Metriken zu berechnen. Eine solche Metrik hilft uns zu verfolgen, wie viele A's, C's, G's und T's wir normalerweise an jeder Position entlang eines DNA-Fragments sehen.
Sobald die Metriken für diesen Datenblock berechnet sind, zeichnen wir die Ergebnisse interaktiv mit Plotly.js und fahren mit dem nächsten Block in der Datei fort. Der Grund für die Verarbeitung der Datei in kleinen Stücken besteht einfach darin, die Benutzererfahrung zu verbessern: Die Verarbeitung der gesamten Datei auf einmal würde zu lange dauern, da FASTQ-Dateien im Allgemeinen Hunderte von Gigabyte groß sind. Wir haben festgestellt, dass eine Blockgröße zwischen 0,5 MB und 1 MB die Anwendung nahtloser machen und Informationen schneller an den Benutzer zurückgeben würde, aber diese Zahl variiert je nach den Details Ihrer Anwendung und dem Umfang der Berechnungen.
Die Architektur unserer ursprünglichen JavaScript-Implementierung war ziemlich einfach:
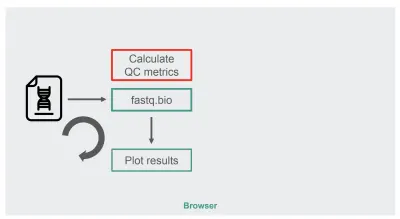
In der roten Box führen wir die String-Manipulationen durch, um die Metriken zu generieren. Diese Box ist der rechenintensivere Teil der Anwendung, was sie natürlich zu einem guten Kandidaten für die Laufzeitoptimierung mit WebAssembly macht.
fastq.bio: Die WebAssembly-Implementierung
Um zu untersuchen, ob wir WebAssembly nutzen könnten, um unsere Webanwendung zu beschleunigen, haben wir nach einem handelsüblichen Tool gesucht, das QC-Metriken für FASTQ-Dateien berechnet. Insbesondere haben wir nach einem in C/C++/Rust geschriebenen Tool gesucht, das für die Portierung auf WebAssembly geeignet ist und das bereits validiert und von der wissenschaftlichen Gemeinschaft als vertrauenswürdig eingestuft wurde.
Nach einiger Recherche entschieden wir uns für seqtk, ein häufig verwendetes, in C geschriebenes Open-Source-Tool, das uns helfen kann, die Qualität von Sequenzierungsdaten zu bewerten (und das allgemeiner zur Bearbeitung dieser Datendateien verwendet wird).
Bevor wir zu WebAssembly kompilieren, überlegen wir uns zunächst, wie wir normalerweise seqtk in eine Binärdatei kompilieren würden, um es auf der Befehlszeile auszuführen. Laut Makefile ist dies die gcc Beschwörung, die Sie benötigen:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lzAndererseits können wir zum Kompilieren von seqtk für WebAssembly die Emscripten-Toolchain verwenden, die Drop-In-Ersatz für vorhandene Build-Tools bietet, um die Arbeit in WebAssembly zu vereinfachen. Wenn Sie Emscripten nicht installiert haben, können Sie ein Docker-Image herunterladen, das wir auf Dockerhub vorbereitet haben und das die benötigten Tools enthält (Sie können es auch von Grund auf neu installieren, aber das dauert normalerweise eine Weile):

$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26 Innerhalb des Containers können wir den emcc Compiler als Ersatz für gcc verwenden:
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1Wie Sie sehen können, sind die Unterschiede zwischen dem Kompilieren in Binärdateien und WebAssembly minimal:
- Anstatt dass die Ausgabe die Binärdatei
seqtkist, bitten wir Emscripten, eine.wasmund eine.js-Datei zu generieren, die die Instanziierung unseres WebAssembly-Moduls handhaben - Um die zlib-Bibliothek zu unterstützen, verwenden wir das Flag
USE_ZLIB; zlib ist so verbreitet, dass es bereits auf WebAssembly portiert wurde, und Emscripten wird es für uns in unser Projekt aufnehmen - Wir aktivieren das virtuelle Dateisystem von Emscripten, das ein POSIX-ähnliches Dateisystem ist (Quellcode hier), außer dass es im RAM innerhalb des Browsers läuft und verschwindet, wenn Sie die Seite aktualisieren (es sei denn, Sie speichern seinen Zustand im Browser mit IndexedDB, aber das ist für einen anderen Artikel).
Warum ein virtuelles Dateisystem? Um dies zu beantworten, vergleichen wir, wie wir seqtk in der Befehlszeile aufrufen würden, mit der Verwendung von JavaScript zum Aufrufen des kompilierten WebAssembly-Moduls:
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"]) Der Zugriff auf ein virtuelles Dateisystem ist leistungsfähig, da wir seqtk nicht neu schreiben müssen, um Zeichenfolgeneingaben anstelle von Dateipfaden zu verarbeiten. Wir können einen Datenblock als Datei data.fastq in das virtuelle Dateisystem einhängen und einfach die main() Funktion von seqtk darauf aufrufen.
Nachdem seqtk zu WebAssembly kompiliert wurde, ist hier die neue fastq.bio-Architektur:
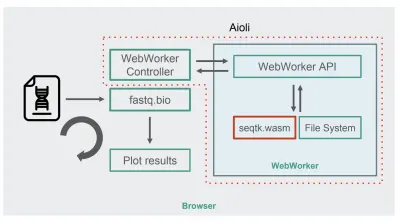
Anstatt die Berechnungen im Haupt-Thread des Browsers auszuführen, verwenden wir, wie im Diagramm gezeigt, WebWorkers, die es uns ermöglichen, unsere Berechnungen in einem Hintergrund-Thread auszuführen und zu vermeiden, dass die Reaktionsfähigkeit des Browsers negativ beeinflusst wird. Insbesondere startet der WebWorker-Controller den Worker und verwaltet die Kommunikation mit dem Haupt-Thread. Auf der Worker-Seite führt eine API die empfangenen Anfragen aus.
Wir können dann den Worker bitten, einen seqtk-Befehl für die gerade gemountete Datei auszuführen. Wenn seqtk die Ausführung beendet hat, sendet der Worker das Ergebnis über ein Promise an den Haupt-Thread zurück. Sobald er die Nachricht erhält, verwendet der Haupt-Thread die resultierende Ausgabe, um die Diagramme zu aktualisieren. Ähnlich wie bei der JavaScript-Version verarbeiten wir die Dateien in Blöcken und aktualisieren die Visualisierungen bei jeder Iteration.
Leistungsoptimierung
Um zu beurteilen, ob die Verwendung von WebAssembly etwas gebracht hat, vergleichen wir die JavaScript- und WebAssembly-Implementierungen anhand der Metrik, wie viele Lesevorgänge wir pro Sekunde verarbeiten können. Wir ignorieren die Zeit, die zum Generieren interaktiver Grafiken benötigt wird, da beide Implementierungen JavaScript für diesen Zweck verwenden.
Im Auslieferungszustand sehen wir bereits eine ~9-fache Beschleunigung:
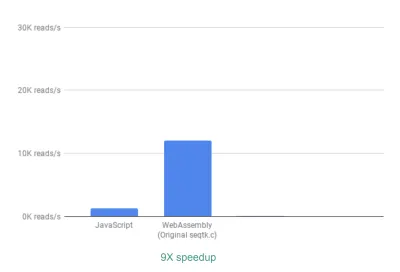
Dies ist bereits sehr gut, da es relativ einfach zu erreichen war (das heißt, sobald Sie WebAssembly verstehen!).
Als Nächstes stellten wir fest, dass seqtk zwar viele allgemein nützliche QC-Metriken ausgibt, viele dieser Metriken jedoch von unserer App nicht wirklich verwendet oder grafisch dargestellt werden. Durch das Entfernen eines Teils der Ausgabe für die Metriken, die wir nicht benötigten, konnten wir eine noch größere Beschleunigung von 13x feststellen:
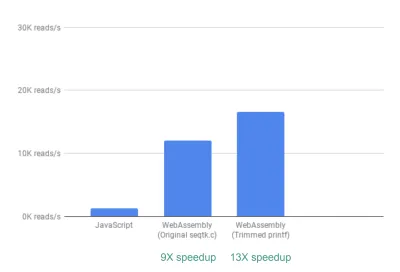
Dies ist wiederum eine große Verbesserung, wenn man bedenkt, wie einfach es zu erreichen war – durch buchstäbliches Auskommentieren von printf-Anweisungen, die nicht benötigt wurden.
Schließlich gibt es noch eine weitere Verbesserung, die wir untersucht haben. Bisher erhält fastq.bio die interessierenden Metriken durch Aufrufen von zwei verschiedenen C-Funktionen, von denen jede einen anderen Satz von Metriken berechnet. Insbesondere gibt eine Funktion Informationen in Form eines Histogramms zurück (dh eine Liste von Werten, die wir in Bereiche einteilen), während die andere Funktion Informationen als Funktion der DNA-Sequenzposition zurückgibt. Leider bedeutet dies, dass derselbe Dateiabschnitt zweimal gelesen wird, was unnötig ist.
Also haben wir den Code für die beiden Funktionen zu einer – wenn auch chaotischen – Funktion zusammengeführt (ohne auch nur mein C auffrischen zu müssen!). Da die beiden Ausgaben eine unterschiedliche Anzahl von Spalten haben, haben wir auf der JavaScript-Seite ein wenig herumgerangelt, um die beiden zu entwirren. Aber es hat sich gelohnt: Dadurch konnten wir eine >20-fache Beschleunigung erreichen!
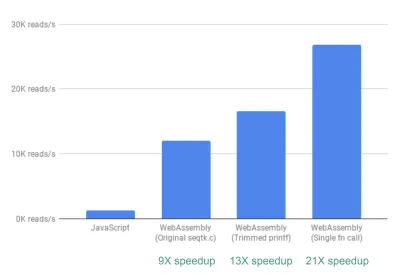
Ein Wort der Warnung
Jetzt wäre ein guter Zeitpunkt für einen Vorbehalt. Erwarten Sie nicht immer eine 20-fache Beschleunigung, wenn Sie WebAssembly verwenden. Möglicherweise erhalten Sie nur eine 2-fache Beschleunigung oder eine 20-prozentige Beschleunigung. Oder es kommt zu einer Verlangsamung, wenn Sie sehr große Dateien in den Arbeitsspeicher laden oder viel Kommunikation zwischen WebAssembly und JavaScript benötigen.
Fazit
Kurz gesagt, wir haben gesehen, dass das Ersetzen langsamer JavaScript-Berechnungen durch Aufrufe von kompiliertem WebAssembly zu erheblichen Beschleunigungen führen kann. Da der für diese Berechnungen benötigte Code bereits in C vorhanden war, hatten wir den zusätzlichen Vorteil, ein vertrauenswürdiges Tool wiederzuverwenden. Wie wir auch angesprochen haben, ist WebAssembly nicht immer das richtige Werkzeug für den Job ( keuch! ), also verwenden Sie es mit Bedacht.
Weiterführende Lektüre
- „Niveau mit WebAssembly“, Robert Aboukhalil
Ein praktischer Leitfaden zum Erstellen von WebAssembly-Anwendungen. - Aioli (auf GitHub)
Ein Framework zum Erstellen schneller Genomik-Webtools. - fastq.bio-Quellcode (auf GitHub)
Ein interaktives Webtool zur Qualitätskontrolle von DNA-Sequenzierungsdaten. - „Eine gekürzte Cartoon-Einführung in WebAssembly“, Lin Clark
