كيف استخدمنا WebAssembly لتسريع تطبيق الويب لدينا بمقدار 20 ضعفًا (دراسة حالة)
نشرت: 2022-03-10إذا لم تكن قد سمعت ، فإليك TL ؛ DR: WebAssembly هي لغة جديدة يتم تشغيلها في المتصفح جنبًا إلى جنب مع JavaScript. نعم هذا صحيح. لم تعد JavaScript هي اللغة الوحيدة التي يتم تشغيلها في المتصفح!
ولكن بخلاف كونه "ليس جافا سكريبت" ، فإن العامل المميز له هو أنه يمكنك ترجمة التعليمات البرمجية من لغات مثل C / C ++ / Rust ( والمزيد! ) إلى WebAssembly وتشغيلها في المتصفح. نظرًا لأن WebAssembly مكتوب بشكل ثابت ، ويستخدم ذاكرة خطية ، ويتم تخزينه في تنسيق ثنائي مضغوط ، فهو سريع جدًا أيضًا ، ويمكن أن يسمح لنا في النهاية بتشغيل التعليمات البرمجية بسرعات "شبه أصلية" ، أي بسرعات تقترب من سرعاتك د الحصول عن طريق تشغيل الثنائي في سطر الأوامر. تعد القدرة على الاستفادة من الأدوات والمكتبات الموجودة لاستخدامها في المستعرض وما يرتبط بها من إمكانات التسريع سببين يجعلان WebAssembly جدًا للويب.
حتى الآن ، تم استخدام WebAssembly لجميع أنواع التطبيقات ، بدءًا من الألعاب (مثل Doom 3) إلى نقل تطبيقات سطح المكتب إلى الويب (مثل Autocad و Figma). يتم استخدامه حتى خارج المتصفح ، على سبيل المثال كلغة فعالة ومرنة للحوسبة بدون خادم.
هذه المقالة عبارة عن دراسة حالة حول استخدام WebAssembly لتسريع أداة الويب لتحليل البيانات. لتحقيق هذه الغاية ، سنأخذ أداة موجودة مكتوبة بلغة C تؤدي نفس العمليات الحسابية ، وتجميعها في WebAssembly ، واستخدامها لاستبدال حسابات JavaScript البطيئة.
ملاحظة : تتعمق هذه المقالة في بعض الموضوعات المتقدمة مثل تجميع كود C ، ولكن لا تقلق إذا لم تكن لديك خبرة في ذلك ؛ ستظل قادرًا على المتابعة والتعرف على ما هو ممكن مع WebAssembly.
خلفية
تطبيق الويب الذي سنعمل معه هو fastq.bio ، وهي أداة ويب تفاعلية توفر للعلماء معاينة سريعة لجودة بيانات تسلسل الحمض النووي الخاصة بهم ؛ التسلسل هو العملية التي نقرأ بها "الحروف" (أي النيوكليوتيدات) في عينة الحمض النووي.
إليك لقطة شاشة للتطبيق أثناء العمل:
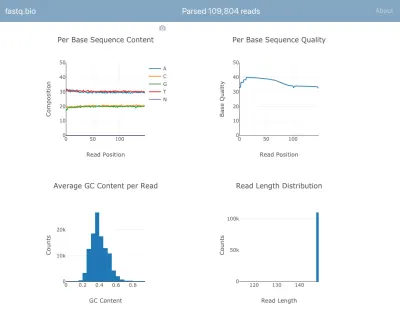
لن ندخل في تفاصيل الحسابات ، ولكن باختصار ، توفر المؤامرات أعلاه للعلماء فكرة عن مدى نجاح التسلسل واستخدامه لتحديد مشكلات جودة البيانات في لمحة.
على الرغم من وجود العشرات من أدوات سطر الأوامر المتاحة لإنشاء تقارير مراقبة الجودة هذه ، فإن هدف fastq.bio هو تقديم معاينة تفاعلية لجودة البيانات دون مغادرة المتصفح. هذا مفيد بشكل خاص للعلماء الذين لا يرتاحون لسطر الأوامر.
المدخلات إلى التطبيق عبارة عن ملف نص عادي يتم إخراجه بواسطة أداة التسلسل ويحتوي على قائمة بتسلسل الحمض النووي ودرجة جودة لكل نوكليوتيد في تسلسل الحمض النووي. يُعرف تنسيق هذا الملف باسم "FASTQ" ، ومن هنا جاء اسم fastq.bio.
إذا كنت مهتمًا بتنسيق FASTQ (ليس ضروريًا لفهم هذه المقالة) ، فراجع صفحة Wikipedia للحصول على FASTQ. (تحذير: تنسيق ملف FASTQ معروف في الحقل لحث راحة الوجه.)
fastq.bio: تنفيذ JavaScript
في الإصدار الأصلي من fastq.bio ، يبدأ المستخدم بتحديد ملف FASTQ من جهاز الكمبيوتر الخاص به. باستخدام كائن File ، يقرأ التطبيق جزءًا صغيرًا من البيانات بدءًا من موضع بايت عشوائي (باستخدام FileReader API). في هذا الجزء من البيانات ، نستخدم JavaScript لإجراء معالجات أساسية للسلسلة وحساب المقاييس ذات الصلة. يساعدنا أحد هذه المقاييس على تتبع عدد A و C و G و T الذي نراه عادةً في كل موضع على طول جزء الحمض النووي.
بمجرد حساب المقاييس لهذا الجزء من البيانات ، نرسم النتائج بشكل تفاعلي مع Plotly.js ، وننتقل إلى الجزء التالي في الملف. سبب معالجة الملف في أجزاء صغيرة هو ببساطة تحسين تجربة المستخدم: معالجة الملف بأكمله دفعة واحدة تستغرق وقتًا طويلاً ، لأن ملفات FASTQ تكون عمومًا بمئات الجيجابايت. وجدنا أن حجم الكتلة بين 0.5 ميغا بايت و 1 ميغا بايت سيجعل التطبيق أكثر سلاسة وسيعيد المعلومات إلى المستخدم بسرعة أكبر ، ولكن هذا الرقم سيختلف اعتمادًا على تفاصيل التطبيق الخاص بك ومدى ثقل الحسابات.
كانت بنية تطبيق JavaScript الأصلي واضحة إلى حد ما:
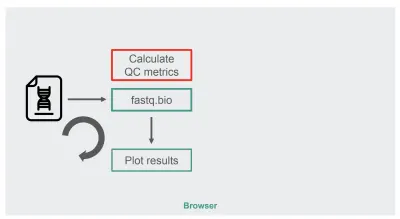
المربع باللون الأحمر هو المكان الذي نقوم فيه بمعالجة السلسلة لإنشاء المقاييس. هذا المربع هو الجزء الأكثر كثافة في الحساب من التطبيق ، مما جعله بطبيعة الحال مرشحًا جيدًا لتحسين وقت التشغيل باستخدام WebAssembly.
fastq.bio: تنفيذ WebAssembly
لاستكشاف ما إذا كان بإمكاننا الاستفادة من WebAssembly لتسريع تطبيق الويب الخاص بنا ، بحثنا عن أداة جاهزة تحسب مقاييس مراقبة الجودة على ملفات FASTQ. على وجه التحديد ، سعينا إلى أداة مكتوبة بلغة C / C ++ / Rust بحيث يمكن نقلها إلى WebAssembly ، والتي تم التحقق من صحتها بالفعل وثقة المجتمع العلمي بها.
بعد إجراء بعض الأبحاث ، قررنا استخدام seqtk ، وهي أداة مفتوحة المصدر شائعة الاستخدام مكتوبة بلغة C يمكن أن تساعدنا في تقييم جودة بيانات التسلسل (وتستخدم بشكل عام لمعالجة ملفات البيانات هذه).
قبل أن نقوم بالتجميع إلى WebAssembly ، دعنا أولاً نفكر في كيفية ترجمة seqtk إلى ثنائي لتشغيله في سطر الأوامر. وفقًا لـ Makefile ، هذا هو gcc الخليجية التي تحتاجها:
# Compile to binary $ gcc seqtk.c \ -o seqtk \ -O2 \ -lm \ -lzمن ناحية أخرى ، لترجمة seqtk إلى WebAssembly ، يمكننا استخدام سلسلة أدوات Emscripten ، والتي توفر بدائل لأدوات الإنشاء الحالية لتسهيل العمل في WebAssembly. إذا لم يكن لديك Emscripten مثبتًا ، فيمكنك تنزيل صورة عامل إرساء قمنا بإعدادها على Dockerhub والتي تحتوي على الأدوات التي ستحتاجها (يمكنك أيضًا تثبيتها من نقطة الصفر ، ولكن هذا يستغرق عادةً بعض الوقت):

$ docker pull robertaboukhalil/emsdk:1.38.26 $ docker run -dt --name wasm-seqtk robertaboukhalil/emsdk:1.38.26 داخل الحاوية ، يمكننا استخدام مترجم emcc كبديل عن دول مجلس التعاون gcc :
# Compile to WebAssembly $ emcc seqtk.c \ -o seqtk.js \ -O2 \ -lm \ -s USE_ZLIB=1 \ -s FORCE_FILESYSTEM=1كما ترى ، الاختلافات بين التحويل البرمجي إلى الثنائي و WebAssembly ضئيلة:
- بدلاً من أن يكون الناتج هو
seqtkالملفات الثنائية ، نطلب من Emscripten إنشاء ملف.wasmو.jsيتولى إنشاء مثيل لوحدة WebAssembly. - لدعم مكتبة zlib ، نستخدم العلم
USE_ZLIB؛ zlib شائع جدًا لدرجة أنه تم نقله بالفعل إلى WebAssembly ، وسوف يقوم Emscripten بتضمينه لنا في مشروعنا - نقوم بتمكين نظام الملفات الظاهري لـ Emscripten ، وهو نظام ملفات يشبه POSIX (رمز المصدر هنا) ، باستثناء أنه يعمل في ذاكرة الوصول العشوائي داخل المتصفح ويختفي عند تحديث الصفحة (إلا إذا قمت بحفظ حالتها في المتصفح باستخدام IndexedDB ، ولكن هذا لمقال آخر).
لماذا نظام الملفات الافتراضي؟ للإجابة على ذلك ، دعنا نقارن كيف يمكننا استدعاء seqtk في سطر الأوامر مقابل استخدام JavaScript لاستدعاء الوحدة النمطية WebAssembly:
# On the command line $ ./seqtk fqchk data.fastq # In the browser console > Module.callMain(["fqchk", "data.fastq"]) يعد الوصول إلى نظام ملفات افتراضي أمرًا قويًا لأنه يعني أنه لا يتعين علينا إعادة كتابة seqtk للتعامل مع مدخلات السلسلة بدلاً من مسارات الملفات. يمكننا تركيب جزء كبير من البيانات على هيئة ملف data.fastq على نظام الملفات الظاهري واستدعاء الدالة main() الخاصة بـ seqtk عليه.
مع تجميع seqtk إلى WebAssembly ، إليك بنية fastq.bio الجديدة:
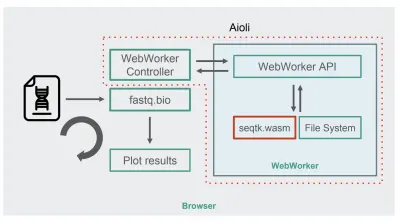
كما هو موضح في الرسم التخطيطي ، بدلاً من تشغيل العمليات الحسابية في الخيط الرئيسي للمتصفح ، فإننا نستخدم WebWorkers ، والتي تتيح لنا تشغيل حساباتنا في سلسلة رسائل في الخلفية ، وتجنب التأثير سلبًا على استجابة المتصفح. على وجه التحديد ، تقوم وحدة تحكم WebWorker بتشغيل العامل وإدارة الاتصال مع مؤشر الترابط الرئيسي. من جانب العامل ، تنفذ API الطلبات التي تتلقاها.
يمكننا بعد ذلك أن نطلب من العامل تشغيل أمر seqtk على الملف الذي قمنا بتثبيته للتو. عند انتهاء seqtk من العمل ، يرسل العامل النتيجة مرة أخرى إلى السلسلة الرئيسية عبر وعد. بمجرد تلقي الرسالة ، يستخدم الخيط الرئيسي الناتج الناتج لتحديث الرسوم البيانية. على غرار إصدار JavaScript ، نقوم بمعالجة الملفات في أجزاء ونقوم بتحديث التصورات في كل تكرار.
تحسين الأداء
لتقييم ما إذا كان استخدام WebAssembly يحقق أي فائدة أم لا ، نقارن تطبيقات JavaScript و WebAssembly باستخدام مقياس عدد القراءات التي يمكننا معالجتها في الثانية. نتجاهل الوقت الذي يستغرقه إنشاء الرسوم البيانية التفاعلية ، لأن كلا التطبيقين يستخدمان JavaScript لهذا الغرض.
من خارج الصندوق ، نرى بالفعل تسريع ~ 9X:
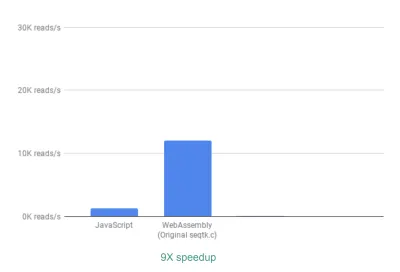
هذا جيد جدًا بالفعل ، نظرًا لأنه كان من السهل نسبيًا تحقيقه (أي بمجرد فهمك لـ WebAssembly!).
بعد ذلك ، لاحظنا أنه على الرغم من أن seqtk ينتج الكثير من مقاييس مراقبة الجودة المفيدة بشكل عام ، فإن العديد من هذه المقاييس لا يستخدمها تطبيقنا أو يرسمها. من خلال إزالة بعض مخرجات المقاييس التي لم نكن بحاجة إليها ، تمكنا من رؤية تسريع أكبر بمقدار 13X:
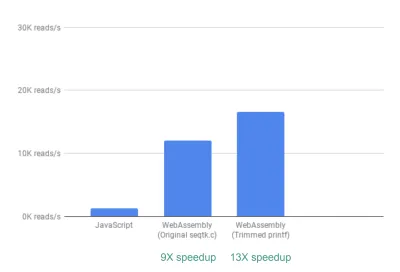
يعد هذا مرة أخرى تحسنًا كبيرًا نظرًا لمدى سهولة تحقيقه — من خلال التعليق حرفيًا على عبارات printf التي لم تكن ضرورية.
أخيرًا ، هناك تحسين آخر نظرنا فيه. حتى الآن ، الطريقة التي يحصل بها fastq.bio على المقاييس ذات الأهمية هي استدعاء وظيفتين مختلفتين من وظائف C ، كل منهما تحسب مجموعة مختلفة من المقاييس. على وجه التحديد ، تقوم إحدى الوظائف بإرجاع المعلومات في شكل مدرج تكراري (أي قائمة القيم التي نقوم بتجميعها في نطاقات) ، بينما تقوم الوظيفة الأخرى بإرجاع المعلومات كدالة لموضع تسلسل الحمض النووي. لسوء الحظ ، هذا يعني أن نفس الجزء من الملف يُقرأ مرتين ، وهو أمر غير ضروري.
لذلك قمنا بدمج كود الوظيفتين في وظيفة واحدة - وإن كانت فوضوية - (دون الحاجة إلى تنظيف C!). نظرًا لأن المخرجين لهما عدد مختلف من الأعمدة ، فقد أجرينا بعض الجدل على جانب JavaScript لفصل الاثنين. ولكن الأمر كان يستحق ذلك: فقد أتاح لنا القيام بذلك تحقيق تسريع يزيد عن 20 ضعفًا!
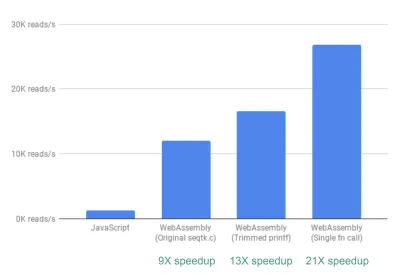
كلمة تحذير
الآن سيكون الوقت المناسب للتحذير. لا تتوقع دائمًا الحصول على تسريع يصل إلى 20 ضعفًا عند استخدام WebAssembly. قد تحصل فقط على تسريع 2X أو 20٪ تسريع. أو قد تتباطأ السرعة إذا قمت بتحميل ملفات كبيرة جدًا في الذاكرة ، أو تطلبت قدرًا كبيرًا من الاتصال بين WebAssembly وجافا سكريبت.
خاتمة
باختصار ، لقد رأينا أن استبدال حسابات JavaScript البطيئة باستدعاءات WebAssembly يمكن أن يؤدي إلى تسريع كبير. نظرًا لأن الكود المطلوب لهذه الحسابات موجود بالفعل في لغة C ، فقد حصلنا على فائدة إضافية تتمثل في إعادة استخدام أداة موثوقة. كما تطرقنا إليه أيضًا ، لن يكون WebAssembly دائمًا الأداة المناسبة للمهمة ( اللحظات! ) ، لذا استخدمه بحكمة.
قراءة متعمقة
- "Level Up With WebAssembly" روبرت أبو خليل
دليل عملي لبناء تطبيقات WebAssembly. - أيولي (على جيثب)
إطار عمل لبناء أدوات ويب سريعة لعلم الجينوم. - كود مصدر fastq.bio (على جيثب)
أداة ويب تفاعلية لمراقبة جودة بيانات تسلسل الحمض النووي. - "مقدمة رسوم متحركة مختصرة حول WebAssembly" لين كلارك
